ゲノムプロファイリングの技術は、過去数十年の間に大きく進歩しました。現在、研究者は、低コストのシークエンシング技術とクロマチン免疫沈降法 (ChIP-seq) などの上流のワークフローと組み合わせることで、短期間で膨大な量のデータを得ることができます。ChIP-seqは、in vivoにおけるタンパク質と標的遺伝子の直接的な結合を測定するためにデザインされたアッセイです。ホルムアルデヒドでタンパク質とDNAをクロスリンクし、細かく断片化したクロマチンを特異的な抗体で免疫沈降します。その後、タンパク質を消化して、残ったDNA断片の配列を決定します。この技術によって、これまでは不可能であった解析ができるようになりました。
タンパク質とDNAの相互作用を直接測定することができれば、遺伝子発現の原因となる証拠を示すことができます。特定のタイプのがんで、ある転写因子が過剰発現することを示すのも1つの方法ですが、これが未知のゲノム領域に物理的に結合することを示すことで、データのレベルが1段階引き上げられます。実際に、直接結合の証拠を要求する学術雑誌は増えています。このため、ゲノムプロファイリングはエピジェネティクスの研究室だけのものではなく、多くの学術機関が独自のシーケンサーコアを持っています。
ごく基本的なところでは、ChIP-seq実験で細胞や組織のヒストン修飾のレベルを検出したり、転写因子の結合をマッピングすることができます。複数のヒストン修飾や転写因子のマッピングなど、複数の項目を一度に調べることができるので、ChIP-seqは非常に用途の広い手法です。例えば、ChIP-seqでH3K4me3とH3K27me3のレベルを測定することで、ヒストンコードから、活性化された遺伝子、不活性化された遺伝子、Bivalent遺伝子を予測することができます。H3K27acレベルを測定することでエンハンサーをマッピングすることができ、クロマチンインスレーターであるCTCFをマッピングすることでクロマチン構造を推測することもできます。Boyerらは2005年、転写因子OCT-4、Sox2、Nanogに対する抗体を用いたChIPアッセイにより、これら全ての因子が共に結合する多くの標的因子を同定しました。このデータから、現在の幹細胞生物学の中核的な概念である、OCT-4、Sox2、Nanogが自己制御ループを形成して多能性と自己複製を担うという仮説が提唱されました。
ChIP-seqアッセイは比較ツールとしても有用です。異なるタイプの細胞や組織、病態をマッピングすることで、根底にある転写回路を特定することができます。この種のデータは、主要ながんドライバーのメカニズムの発見に寄与しています。Zanconatoらは2015年、乳がん細胞の発がん性転写因子YAP + TAZのマッピングを行い、エンハンサーへの結合から、YAP/TAZの未知の機能を発見しました。さらに、野生型細胞とsiRNA処理した細胞を比較し、YAP/TAZによる腫瘍の成長とAP-1の間に重要な関連性があることを明らかにしました。
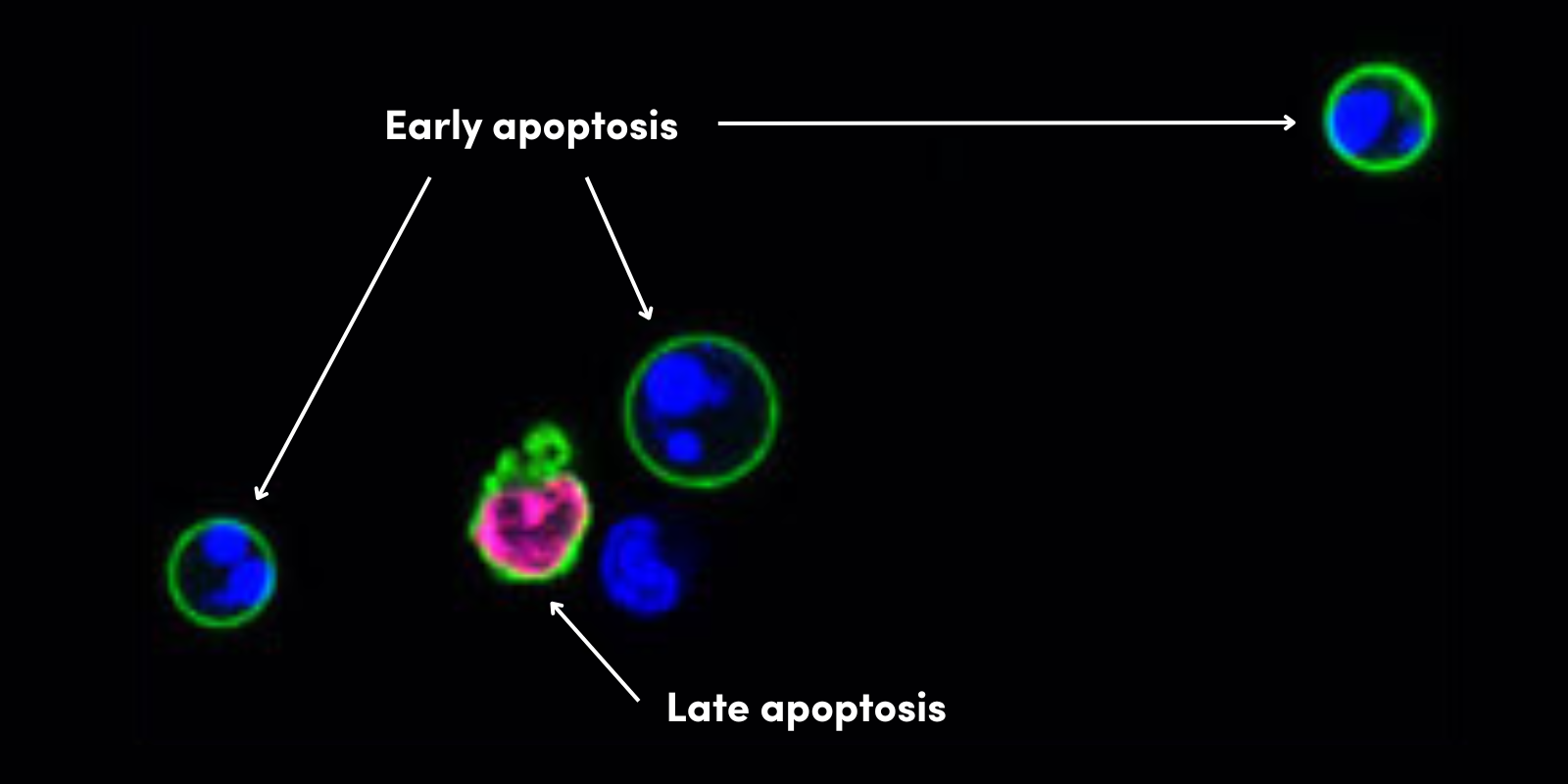
しかし、ChIPやChIP-seqにも限界はあります。多くの場合、大量の細胞が必要で、患者サンプルや初代培養細胞の解析は困難です。免疫沈降のステップの効率が非常に悪いことや、クロマチンを予めクロスリンクして断片化する必要があることがこの原因です。Steve Henikoff博士の研究室で開発されたCUT&RUN (Cleavage Under Target & Release Using Nuclease) と呼ばれる新技術は、標的抗体を基盤としたin situのゲノム断片化を利用して、ChIP-seq実験と同等の機能を果たします。このアッセイにクロスリンクや免疫沈降のステップは不要で、必要な細胞数が大幅に低減され、シーケンスにおけるバックグラウンドも低下します。
必要な細胞数が少ないことから、研究や医療に大きな進歩をもたらす可能性があります。幹細胞生物学者にとって、iPS細胞のゲノムプロファイリングは、ChIP-seqに必要な量の細胞を確保するために膨大な時間と労力が求められるため、非常に困難でした。免疫学の分野では、T細胞の疲弊にエピジェネティクスが及ぼす影響について、長らく疑問が残っています。CUT&RUNを利用することで、供給の限られた、単離した免疫細胞を最大限に活用し、タイプの異なる細胞間でゲノムの状況を比較する研究が可能になるかも知れません。個々の患者の腫瘍のマッピングにより、主要な転写因子の活性を決定し、個別化医療に貢献する可能性もあります。今後の展望は明るく、実験に必要なのは研究者の想像力 (と、協力が得られるバイオインフォマティクス部門) だけです。
参考文献:
-Strahl BD, Allis CD. The language of covalent histone modifications. Nature. 2000;403(6765):41-45. doi:10.1038/47412
-Bernstein BE, Mikkelsen TS, Xie X, et al. A bivalent chromatin structure marks key developmental genes in embryonic stem cells. Cell. 2006;125(2):315-326. doi:10.1016/j.cell.2006.02.041
-Boyer LA, Lee TI, Cole MF, et al. Core transcriptional regulatory circuitry in human embryonic stem cells. Cell. 2005;122(6):947-956. doi:10.1016/j.cell.2005.08.020
-Chen XB、Dou Z、Xu G、He XY、Yang YXA kind of universal quantum secret sharing protocol. Sci Rep. 2017;7:39845. Published 2017 Jan 12. doi:10.1038/srep39845