ChIP-SeqやCUT&RUN、最新の手法であるCUT&Tagなどを用いたクロマチンプロファイリングは、疾患の発症機構あるいはエピジェネティックな変化の調節を新規の治療法の開発にどのように活用できるかの理解に大きく貢献してきました。これらの3種類の技術のいずれかの手法でDNAライブラリーを調製した後に、NanoDropやQubitシステムなどのプラットフォームでライブラリーを定量し、BioanalyzerやTapeStationシステムなどの装置を用いて品質の確認を行い、次世代シーケンシング (NGS) で解析します。しかし、CUT&Tag DNAライブラリーの濃度が低い場合や、BioanalyzerやTapeStationシステムで作成されるシグナルが低い場合はどうすれば良いでしょうか?このようなDNAライブラリーでも、シーケンシングを成功させることができる方法を紹介します。
DNAライブラリーの定量方法はどれも同じ結果を示すとは限らない
Bioanalyzerまたは TapeStationの装置は、調製したDNAサンプルやRNAサンプルの品質管理によく用いられます。解析の前に、Thermo Fischer Scientific社のNanodrop装置、Qiagen社のQIAxpertシステム、Thermo Fisher Scientific社のQubit蛍光測定システムまたはPicoGreenアッセイを用いてライブラリーの濃度を決定できます。しかし、増幅されたCUT&Tag DNAライブラリーの収量の測定値は、用いる手法によって様々です。CSTの科学者は、異なる手法で同じDNAライブラリーを測定し、以下のCUT&Tag DNAライブラリーの収量に関するガイドラインを作成しました。収量が低い場合でも、品質の高いNGSデータが取得できることを覚えておいてください。
| 方法 | 標的 | 予想される濃度 | 推奨される対処法 |
| NanoDropまたはQIAxpertシステム | ヒストン | 10 - 20 ng/µL | DNAライブラリーの濃度が3 ng/µLを超える場合は、BioAnalyzer またはTapeStationシステムを用いたQC (品質管理) において、ライブラリーのシグナルが低いまたは見当たらなくても、NGS解析に進むことを推奨します。ライブラリーの濃度が3 ng/µLより少ない場合は、CSTのトラブルシューティングガイドを参照してください |
| コファクターまたは 転写因子 |
5 - 12 ng/µL | ||
| Qubit蛍光測定システムまたはPicoGreenアッセイ | ヒストン | 3 - 10 ng/µL | BioAnalyzerまたはTapeStationシステムで測定するには濃度が低すぎる可能性があります。Tri-Methyl-Histone H3 (Lys4) などのポジティブコントロールで期待通りのライブラリーの収量が得られる、またはBioanalyzerまたはTapeStationでピークが作成される、もしくはqPCRを用いた CUT&Tag DNAライブラリーの品質管理の際に良好なS/N比が得られる場合は、ライブラリーをNGSで解析することを推奨します。 |
| コファクターまたは 転写因子 |
<1 ng/µL |
CUT&Tagのよくある質問のページを参照して、サポートデータや、収量が異なるサンプルを混合する際の注意事項をご覧ください。
DNAライブラリーの品質管理
BioanalyzerまたはTapeStationシステムにより、サンプルの濃度や平均した断片サイズの範囲、サンプルの純度が分かります。標的が転写因子などの場合は、細胞内での存在量が少ないまたはサンプルとなる細胞数が少ないといった理由から、BioanalyzerまたはTapeStation装置が示すライブラリーの収量が低くなることがあります。このような場合、NGSに進むべきかどうかを判断することは難しく、ChIP-seqやCUT&RUNのDNAライブラリーであれば、NGSに進むのを断念する研究者も多いでしょう。しかし、CUT&Tagの基準となる閾値はより低くなっているため、次のような疑問が浮かびます。BioanalyzerやTapeStationシステムでのシグナルが弱くても、CUT&Tag DNAライブラリーのシーケンスは正常に行えるのでしょうか?
CUT&Tagでは、BioanalyzerやTapeStationでのシグナルが弱くても質の高いNGSデータを取得可能
標的の存在量が低いまたは細胞数が少ない場合は、プロトコールを成功に導くために、Tri-Methyl-Histone H3 (Lys4) などのポジティブコントロールを用いて調製したDNAライブラリーと共に解析することを推奨します1。ポジティブコントロールのシグナルが強く、標的となるライブラリーのシグナルが弱い場合は、そのCUT&Tag DNAライブラリーを用いて正常にシーケンシングを行い、重要なタンパク質-DNA相互作用データを取得できます (図1)。そのため、調製したCUT&Tag DNAライブラリーが、BioanalyzerまたはTapeStationシステムでのプロファイリングでピークが非常に弱いまたはピークがみられない場合も、シーケンシングに進むことを推奨します。
Bioanalyzerシステムで弱いシグナルを示した3種類のDNAライブラリーから得られた、正確なTCF4/TCF7L2のシーケンシングデータ
.jpg?width=780&height=177&name=23-ETC-03854%20CUT%26Tag%20Bioanalyzer_Blog%20Image1%20(1).jpg)
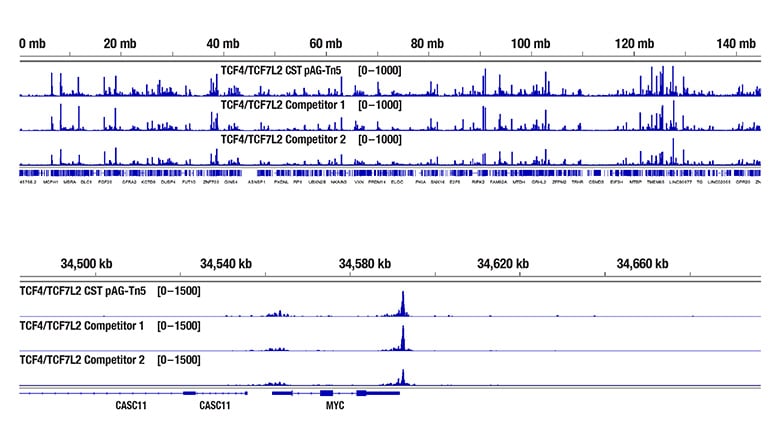
図1. HCT 116細胞、TCF4/TCF7L2 (C48H11) Rabbit mAb #2569、他社製品を含むいくつかのpAG-Tn5を用いてCUT&Tagを行いました。使用した各酵素の量は、メーカーの推奨量に基づいています。DNAライブラリーは、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を用いて調製しました。上の図は、Bioanalyzerシステムで得られた、3種類のCUT&Tag DNAライブラリーのプロファイリングの結果を示しています。
下の図は、Bioanalyzerシステムで解析したものと同じDNAライブラリーをシーケンシングし、得られたNGSトラックを示しています。NGSの結果から、8番染色体全体への結合 (上) およびTCF4/TCF7L2の既知の標的遺伝子であるMYCへの結合 (下) において、同等の結果が得られたことが分かります。
qPCR:代替となるCUT&Tag DNAライブラリーの品質管理方法
BioanalyzerまたはTapeStationシステムで得られたシグナルが低いCUT&Tag DNAライブラリーのシーケンスに懸念が残る場合は、NGSの前に既知のポジティブおよびネガティブ遺伝子座を用いたDNAライブラリーのqPCRを行い、クロマチン断片の濃縮を評価して品質管理する方法があります。qPCRは、ライブラリー調製前のCUT&Tag DNAではなく、CUT&Tag DNAライブラリーに対して実行する必要があることにご注意ください。CUT&Tagアッセイの最終段階で核膜が破壊されるため、サンプル中にはタグ付けされた小さなDNAと大きなDNAの両方が存在します。そのため、ライブラリー増幅前のCUT&Tag DNAは、qPCR解析に適していません。タグ付けされたDNAは、DNAライブラリーのPCR増幅中に選択的に濃縮されるため、ゲノムDNAは効果的に希釈されます。これにより、CUT&Tag DNAライブラリーは、qPCR解析に適したものになります。qPCRで得られるS/N比が良好であれば、CUT&Tag DNAライブラリーもシーケンスにも成功できることを示す強力な指標となります。
CUT&Tag DNAライブラリーはS/N比が大幅に高いqPCRデータを生成します
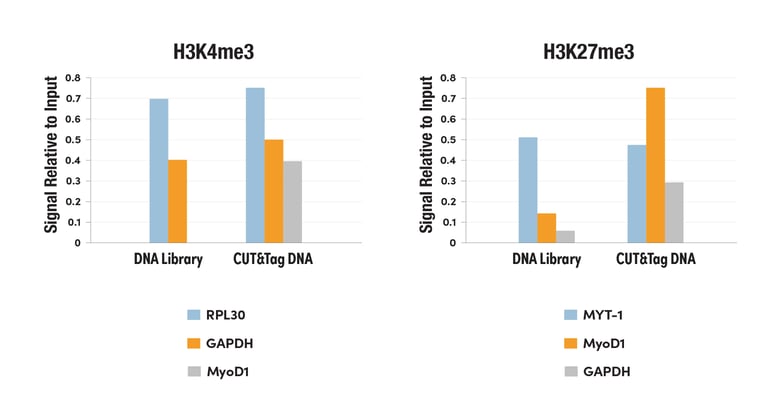
図2. HeLa細胞、CUT&Tag Assay Kit #77552、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751を用いてCUT&Tagを行いました。DNAライブラリーは、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を用いて調製しました。H3K4me3の既知の結合部位に対するポジティブプライマーセットとしてSimpleChIP® Human RPL30 Exon 3 Primers #7014とHuman GAPDH Exon 1 Primersを、H3K4me3の非結合部位に対するネガティブプライマーセットとしてSimpleChIP Human MyoD1 Exon 1 Primers #4490を用いて、濃縮したDNAをリアルタイムPCRで定量しました。各サンプル中の免疫沈降されたDNAの量は、インプットクロマチンの総量を1とした相対的なシグナルとして示しています。
結論になりますが、BioanalyzerやTapeStationシステムで得られたシグナルが低いからといって、CUT&TagライブラリーDNAのシーケンスをあきらめないでください。きっと質の高い結果が得られます。
参考文献:
-
Kaya-Okur HS, Wu SJ, Codomo CA, et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nat Commun. 2019;10(1):1930. Published 2019 Apr 29 doi:10.1038/s41467-019-09982-5
C-03854