20年前、CSTの科学者は、細胞内シグナル伝達を調査するためのより堅牢なプロテオミクス研究に必要なツールを考えていた際に、ある革新的な問いに行き当たりました:もしも、抗体が隣接するアミノ酸配列に関係なく翻訳後修飾 (PTM) に結合することができたらどうだろうか?
この問いと、この問いから生まれたCSTが開発した手法は、PTMScan®製品とCST®プロテオミクスサービスの礎となっています。これらにより、液体クロマトグラフィータンデム質量分析 (LC-MS/MS) を用いて、数千もの新たなPTM部位を迅速に特定・定量化することができるようになるため、プロテオミクス解析を簡便化することができます。
本ブログでは、この画期的な抗体の作製方法や、この技術を用いた革新的な研究をいくつかご紹介します。
プロテオミクス研究の弱点の発見
2000年初頭、CSTの科学者は多くの研究者が直面する課題に取り組んでいました。当時のPTM解析手法は、感度や特異性、処理能力が不十分であり、サンプル中により多く存在するタンパク質を重点的に解析する傾向にありました。つまり、発現が低いタンパク質を含むシグナル伝達経路は、疾患の進行に重要であるにも関わらず、見逃されていた可能性があるのです。
CSTの科学者は、医薬品の開発や変異の解析、キナーゼのプロファイリングの研究用に抗体開発のプロセスを転換し、現在も様々な発見を牽引している新しいPTM検出方法を開発しました。それが、PTMScanです。
PTMを検出する新たな手法:PTMScanとは?
PTMScanは、独自の抗体開発アプローチを採用しており、1回のLC-MS/MSで数千もの新たなPTM部位を検出できます。この技術は、リン酸化やユビキチン化、アセチル化、その他の広範なPTM研究において、すでに成功を収めています。
PTMScanの開発時、CSTの科学者は、がん細胞内の異常なチロシンキナーゼの活性を特定する方法を探していました1。活性化したチロシンキナーゼは、がんのシグナル伝達のドライバーであることが知られていますが、シグナル伝達経路が複雑かつチロシンリン酸化レベルが低いため (トータルリン酸化プロテオーム量の1%未満)、特異的な基質の特定は困難です。当時、既知のチロシンリン酸化イベントに対する抗体の開発は可能でしたが、新たな部位に関しては開発は不可能でした。
新規PTM部位検出用の抗体の開発
まずは、科学者が一般的にどのように抗体を作製しているかを考えてみましょう。一般的に抗体は、抗原に特異的な抗体を作製するために、ラビットの免疫系にペプチド抗原を投与します。これにより、単一のペプチド配列、または標的配列内に特異的なアミノ酸残基のPTM部位を含むタンパク質に親和性をもつ、部位特異的なPTM抗体が得られます。以下の例では、単一のリン酸化ペプチドを抗原として用いており、標的とするリン酸化チロシン残基をもつペプチド配列にのみ反応する、特異的な抗体を開発することができます。
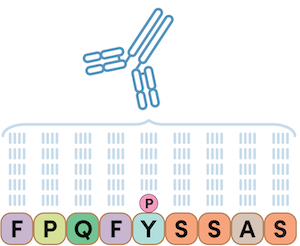
図1:リン酸化ペプチド抗原をラビットに投与すると、リン酸化ペプチド配列に特異的な抗体を作製することができます。上の例は、部位特異的なリン酸化チロシン抗体を示しています。PTMScan Direct関連製品は、この検出技術をベースにしています。
PTMScanは、単一のリン酸化ペプチド配列に特異的な抗体ではなく、抗体の開発に用いられるペプチドライブラリーをベースとするアプローチを用いています。これは、リン酸化されたアミノ酸残基 (またはその他のPTM残基) だけに親和性をもつ特異的な抗体を作製するアプローチです。この戦略は、その他の翻訳後修飾された残基 (アセチルリシンやメチルアルギニンなど) に特異的な抗体の作製にも応用することができます。このアプローチでは、リン酸化チロシン特異的抗体を得るために、リン酸化チロシン部位を固定し、その他の隣接する位置はランダムなアミノ酸のペプチドライブラリーを用いてラビットを免疫します1。
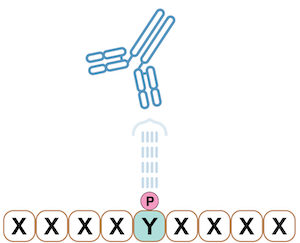 図2:リン酸化チロシン残基を固定し、隣接する位置 (Xの印) はランダムなアミノ酸のペプチドライブラリーの例。上の例は、リン酸化チロシン特異的抗体を示しています。この技術は、PTMScan Discovery製品の作製に用いられています。
図2:リン酸化チロシン残基を固定し、隣接する位置 (Xの印) はランダムなアミノ酸のペプチドライブラリーの例。上の例は、リン酸化チロシン特異的抗体を示しています。この技術は、PTMScan Discovery製品の作製に用いられています。
あらゆるリン酸化チロシン部位に特異的な抗体を用いることにより、消化後の複雑な混合物からリン酸化チロシンをもつすべてのペプチドを免疫沈降し、その後LC-MS/MSを用いて特定することができます。
さらに、PTMに特異的な抗体を作製する手法は改良され、リン酸化された基質モチーフを調査できるようになりました。このアプローチでは、リン酸化セリン部位と保存された基質モチーフを固定し、残りの隣接する位置はランダムなアミノ酸となるペプチドライブラリーを用いてラビットを免疫します1。
下の例は、アルギニン残基がリン酸化セリンの3つ前に固定されているモチーフ抗体を示しています。ペプチドライブラリーでは、これらの2つの残基は固定されており、これらの残基の周辺のアミノ酸はランダムになっています。このモチーフ抗体は、セリン残基がリン酸化された時のみ、あらゆるタンパク質のRXXpS配列を認識します。これはAKTシグナル伝達経路の主要な部分を担うAKTの基質モチーフに特異的な配列です。
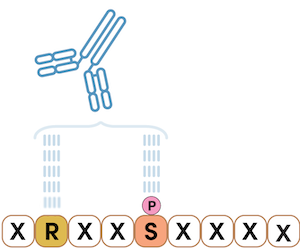
図3:2つの位置のみが固定された基質モチーフRXXpS (Xはいずれかのアミノ酸)。この例では、アルギニン残基がリン酸化セリンの3つ前の位置に固定されています。モチーフ抗体はPTMScan Discoveryでも用いられています。
この手法により、標的である修飾モチーフに非常に特異的でありながら、様々な一次アミノ酸配列に対して幅広く反応する抗体を開発することができます。
ボトムアッププロテオミクス用のCST製品
CSTは、上述した抗体開発方法を用いて、標的研究と初期探索用のプロテオミクス解析の簡便化に役立つ一連のPTMScan製品を提供しています。これにより、モデルシステムにおいてシグナル伝達を駆動する重要なPTMを効率よく特定できます。
PTMScan® Direct
標的研究では、PTMScan Directは創薬の初期段階で標的やオフターゲット効果を特定し、創薬パイプラインのリスクを軽減します。この技術は、解析対象となるシグナル伝達経路における、標的タンパク質上の既知のPTMの経路を中心としたスクリーニングや標的化を可能とし、薬物処理に対する細胞内応答のモニタリング、または疾患状態のドライバーの特定に用いることができます。PTMScan Directは、シグナル伝達経路や細胞内区画における特異的かつ代表的なタンパク質がもたらす、数千ものシグナルノードの特定や定量を可能にします。
PTMScan® Multi-Pathway Enrichment Kitは、4,000を超えるユニークな部位と、機能がすでに分かっている80を超える主要なタンパク質を1回の実験でプロファイリングすることができます。
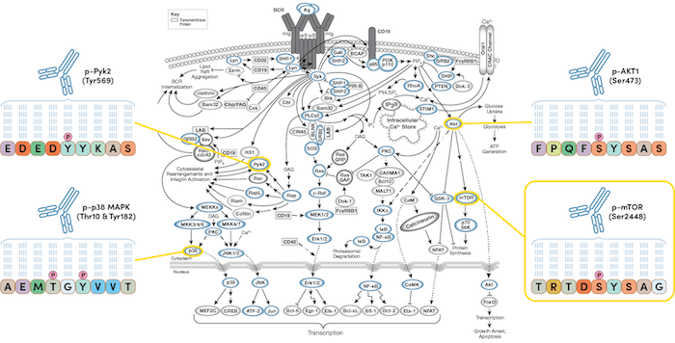
図4PTMScan Directは、特定のシグナル伝達経路のグループにおける標タンパク質標上の既知のPTMの調査に用いることができます。
PTMScan Discovery
探索研究でPTMScan Discoveryを用いる場合は、プロテオーム全体を調査して目的のPTMの特定および定量を行います (図5)。
PTMScan Motif Antibodyキットを用いて行うPTMScan Discoveryは、実験条件への反応、または疾患の進行 (もしくは表現型) の原因となるタンパク質の主要な部位を解明するために、コントロール状態と摂動状態の間で調節されるPTMを、プロテオーム全体を通して特定します。
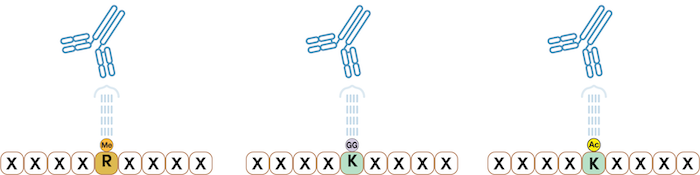 図5:PTMScan Discovery製品は、サンプル中の特定のPTMを検出することができます。左:メチルアルギニン特異的抗体。中央:ユビキチン/SUMO-Remnant特異的抗体。右:アセチル-リシン特異的抗体。
図5:PTMScan Discovery製品は、サンプル中の特定のPTMを検出することができます。左:メチルアルギニン特異的抗体。中央:ユビキチン/SUMO-Remnant特異的抗体。右:アセチル-リシン特異的抗体。
PTMScan HS:より少ないサンプルで行うプロテオミクス探索
PTMScan試薬の感度と特異性を高めるため、CSTの科学者は専門知識を駆使して、磁気ビーズを標識したPTMScan抗体を作製しました。これにより、酸による溶出を行う際に、ビーズから外れてしまう抗体タンパク質を大幅に削減できます。また、この新しいPTMScan製品であるPTMScan HS for High Sensitivityには、バックグラウンドでのペプチド結合の低減に役立つ、新しい結合バッファーおよび洗浄バッファーが含まれています。特に重要な点は、これらのキットは必要とするサンプル量が少なく、磁気ビーズを用いるため実験台での作業量が大幅に減り、作業の自動化が可能であることです2。
CSTのプロテオミクスサービス:PTMScanサービス
質量分析装置を利用することができないけれども、探索研究またはPTMを標的とした研究を行いたい研究者に対し、CSTプロテオミクスサービスチームは研究室の延長的な役割を果たし、上述の技術を用いてサンプル内のタンパク質またはPTM部位の定性的・定量的なプロファイリングを行います。CSTの科学者は、お客様と相談しながらプロジェクトの初期の計画から包括的なデータセットの提供までを行い、研究の目標達成をお約束します。
PTMScan技術を用いた発見
PTMScan技術は、開発されてすぐに肺がん3や卵巣がん4、急性骨髄性白血病5などにおけるチロシンリン酸化の新たな活性化因子の発見に役立ちました。また、PTMScanキットの製品化により、世界中の科学者によるチロシンリン酸化の新たな部位やその上流のキナーゼの特定が推進されました。現在では、がん専門医が、がんの増殖を促進するキナーゼに対する既知の阻害剤を、患者に適合させるための情報を得ることができます。
CSTの科学者は、続けてタンパク質上のユビキチン化およびSUMO化を特定するための、リシンアシル化 (アセチル化、ブチリル化、プロピオニル化、マロニル化) に対するPTMScan抗体と試薬を開発しました。トリプシン消化により生成される、ユビキチンレムナントモチーフに対する固有の抗体を用いたPTMScan研究は、数千に及ぶタンパク質上の、数万ものユビキチン化部位の特定と発見につながりました。さらに、これらの研究から、多発性骨髄腫の治療におけるLenalidomideの作用機構6や、多能性や細胞のリプログラミングの調節7、浸潤性の高い表現型をもつヘルペスウイルスが必要とするユビキチン化8、関節炎におけるユビキチンリガーゼの基質9などが解明されました。
より最近では、骨髄腫におけるアルギニンメチルトランスフェラーゼ阻害剤の標的を研究していた科学者が、mRNAのスプライシング因子においてメチル化が大幅に減少していることを発見しました10。メチルトランスフェラーゼの阻害剤はすでに、一部のがん細胞の成長を遅らせることが報告されています。また、アルギニンのメチル化部位を濃縮するために開発されたPTMScanの新たな試薬を導入した研究室は、がん細胞を攻撃するための新しいわずかな隙を発見しました。これらのスプライシング因子は、mRNAを適切にスプライシングするために、PRMT5による主要なアルギニン残基のメチル化を必要とします。誤ったスプライシングを受けるmRNAの中には、細胞周期やp53の調節に重要なMDM4などのタンパク質が含まれます。これらの研究や、PTMScanを用いた類似の実験により、がん細胞の成長を促進する重要なタンパク質が特定されました。
PhosphoSite:PTMの無料オンラインデータベース
CSTの科学者は、PTMScanの開発と同時に、厳選されたタンパク質のリン酸化を検索できる無料オンラインデータベースPhosphoSitePlus.orgを立ち上げました。Phosphositeは、投稿されたタンパク質リン酸化のデータと弊社の研究結果を組み合わせた、PTMのオンラインデータベースです11,12。
PhosphoSitePlusはさらに拡張し、タンパク質のユビキチン化、アセチル化、リシンメチル化、アルギニンメチル化、およびN-グリコシル化が加わり、世界中の研究者が数百、数千ものPTMに関する情報にアクセスすることができます。
20年にわたる革新的プロテオミクス研究
世界中の科学者が発表した、PTMScanを採用した査読付きの論文は100報を超えています。PTMScanは、新たなPTM特異的抗体、最先端の質量分析、およびバイオインフォマティクスの3つの技術の相乗効果により、その力を発揮します。この相乗効果により、1回の実験でPTMの数千もの部位の特定と相対定量が可能となります。個別化医療の未来は、異常な増殖の原因を分子レベルで特定することにかかっています。そのため、20年経った現在でも、PTMペプチドの濃縮というバイアスの少ないアプローチをもつPTMScan技術は、探索用ツールとして比類のない力を誇っています。
CSTのプロテオミクスについて詳しく知る
- PTMScanPathway-Based EnrichmentキットやPTMScan Motif AntibodyキットなどのPTMScanキットは、CSTのウェブサイトからご購入いただけます
- CSTは、PTMScanを用いたプロテオミクス解析サービスも提供しています
- 関連ブログ:3倍のデータ、3倍の知見を取得可能:DIAプロテオミクスとDDAプロテオミクスの違い
- 関連ブログ:プロテオミクス実験における再現性:PTMScanでコントロールペプチドを使用する
参考文献
- Rush J, Moritz A, Lee KA, et al. Immunoaffinity profiling of tyrosine phosphorylation in cancer cells. Nat Biotechnol. 2005;23(1):94-101. doi:10.1038/nbt1046
- Rivera KD, Olive ME, Bergstrom EJ, et al. Automating UbiFast for High-throughput and Multiplexed Ubiquitin Enrichment. Mol Cell Proteomics. 2021;20:100154. doi:10.1016/j.mcpro.2021.100154
- Rikova K, Guo A, Zeng Q, et al. Global survey of phosphotyrosine signaling identifies oncogenic kinases in lung cancer. Cell. 2007;131(6):1190-1203. doi:10.1016/j.cell.2007.11.025
- Ren H, Zhi-Ping T, Zhu X, Crosby K, Haack H, Ren JM, Beausoleil S, Mortiz A, Innocenti G, Rush J, Zhou X, Gu TL, Yang YF, Comb M. Identification of Anaplastic Lymphoma Kinase as a Potential Therapeutic Target in Ovarian Cancer. Cancer Res (2012) 72 (13) 3312-3323
- Gu TL, Goss VL, Reeves C, et al. Phosphotyrosine profiling identifies the KG-1 cell line as a model for the study of FGFR1 fusions in acute myeloid leukemia. Blood. 2006;108(13):4202-4204. doi:10.1182/blood-2006-06-026666
- Krönke J, Udeshi ND, Narla A, et al. Lenalidomide causes selective degradation of IKZF1 and IKZF3 in multiple myeloma cells. Science. 2014;343(6168):301-305. doi:10.1126/science.1244851
- Buckley SM, Aranda-Orgilles B, Strikoudis A, et al. Regulation of pluripotency and cellular reprogramming by the ubiquitin-proteasome system. Cell Stem Cell. 2012;11(6):783-798. doi:10.1016/j.stem.2012.09.011
- Huffmaster NJ, Sollars PJ, Richards AL, Pickard GE, Smith GA. Dynamic ubiquitination drives herpesvirus neuroinvasion. Proc Natl Acad Sci U S A. 2015;112(41):12818-12823. doi:10.1073/pnas.1512559112
- Lee KA, Hammerle LP, Andrews PS, et al. Ubiquitin ligase substrate identification through quantitative proteomics at both the protein and peptide levels. J Biol Chem. 2011;286(48):41530-41538. doi:10.1074/jbc.M111.248856
- Musiani D, Bok J, Massignani E, et al. Proteomics profiling of arginine methylation defines PRMT5 substrate specificity. Sci Signal. 2019;12(575):eaat8388. Published 2019 Apr 2. doi:10.1126/scisignal.aat8388
- Hornbeck PV, Zhang B, Murray B, Kornhauser JM, Latham V, Skrzypek E. PhosphoSitePlus, 2014: mutations, PTMs and recalibrations. Nucleic Acids Res. 2015;43(Database issue):D512-D520. doi:10.1093/nar/gku1267
- Johnson JL, Yaron TM, Huntsman EM, et al. An atlas of substrate specificities for the human serine/threonine kinome. Nature. 2023;613(7945):759-766. doi:10.1038/s41586-022-05575-3
CST and Cell Signaling Technology are trademarks or registered trademarks of Cell Signaling Technology, Inc. All other trademarks are the property of their respective owners. Visit cellsignal.com/trademarks for more information. 23-BPA-73950
23-BPA-73950